Laboratorio di
Microbiologia Generale
a cura di Gabriele Giliberti

Trasformazione batterica
Alcune specie batteriche possono acquisire DNA estraneo
dall’ambiente e vengono dette per questo motivo “naturalmente competenti”. La competenza, ossia la capacitŕ di
acquisire DNA dall’esterno, dipende dalla secrezione all’esterno della cellula
di una molecola di natura polipeptidica, detta fattore di competenza, che
induce nella cellula batterica la sintesi di specifici recettori di membrana. Questi
recettori legano il DNA e lo trasportano nel citoplasma dove, se esistono
regioni di omologia tra il DNA estraneo e quello cellulare, avviene un evento
di ricombinazione che determina l’integrazione del DNA estraneo sul cromosoma e
la sua eventuale espressione nella cellula ospite. Tutti i batteri tuttavia
hanno la capacitŕ di assumere DNA “nudo” dall’ambiente, ma č necessario indurre
in laboratorio uno stato di competenza
artificiale. Ciň č reso possibile da una temporanea permeabilitŕ della
membrana cellulare al DNA, ottenuta con metodi chimici (esponendo le cellule ad
elevate concentrazioni di ioni metallici come ad es. ioni Ca++) o
fisici (esponendo le cellule ad impulsi di corrente ad alto voltaggio per tempi
molto brevi come nell’elettroporazione). Tali metodi di trasformazione
artificiale sono particolarmente efficaci con molecole di DNA circolare e sono
comunemente utilizzati per introdurre molecole di DNA ricombinante in cellule
batteriche. Infatti, quando si vuole far esprimere un frammento di DNA di
interesse esso viene inserito in un vettore di DNA in grado di replicarsi
selettivamente in un altro organismo piů semplice come il batterio Escherichia coli, permettendone cosě
l’amplificazione e l’espressione. La trasformazione
di E. coli č resa possibile dall’effetto
combinato del CaCl2 e del calore che rendono la membrana
temporaneamente “permeabile” all’ingresso di DNA plasmidico (pUC19), con meccanismi molecolari che rimangono
ancora oggi parzialmente sconosciuti.
Protocollo sperimentale
ü
Effettuare una lettura
spettrofotometrica di una coltura di Escherichia
coli (TOP10) in crescita ogni 30 minuti, finché la coltura non arriva in
fase medio-logaritmica.
ü
Prelevanre 1 ml dalla coltura e
leggere il valore di assorbanza a 600nm, contro un “bianco” contenente solo LB.
ü
Quando la coltura in crescita
raggiunge una OD600 di 0.4, prelevare sterilmente 1,5 ml dalla
coltura e mettere in ghiaccio per 10 minuti.
ü
Centrifugare per 5 minuti a 4000rpm
a 4°C.
ü
Eliminare il sopranatante
sterilmente e mettere in ghiaccio il pellet cellulare.
ü
Risospendere il pellet in 1,5 ml di
una soluzione 0.1M di CaCl2 fredda.
ü
Lasciare in ghiaccio per 10 minuti.
ü
Centrifugare nuovamente per 5 minuti
a 4000rpm a 4°C.
ü
Eliminare il sopranatante
sterilmente e mettere in ghiaccio il pellet cellulare.
ü
Risospendere il pellet in 200µl
della soluzione 0.1M di CaCl2 fredda e mantenere in ghiaccio.
ü
Marcare due provette sterili nel
seguente modo: TOP10 + pUC19 (trasformazione); TOP10 (controllo).
ü
Prelevare 100µl di cellule
competenti e introdurli nelle due provette.
ü
Aggiungere 5µl di una soluzione 10
pg/µl del plasmide pUC19 nella provetta TOP10 + pUC19 e mescolare con
delicatezza.
ü
Lasciare entrambe le provette in
ghiaccio per 15 minuti.
ü
Sottoporre le cellule ad uno shock
termico, ponendo le provette in un bagnetto a 42°C e lasciarle per 90 secondi
[Fig. 1].
ü
Rimettere immediatamente le provette
in ghiaccio per un 1 minuto.
ü
Aggiungere 900µl di terreno LB in
ciascuna provetta.
ü
Incubare le provette per 30 minuti
in un agitatore a 37°C.
ü
Preparare una piastra di terreno
solido LB agar, versando circa 25ml di terreno LB agar, e lasciare
solidificare.
ü
Preparare 3 piastre di terreno
solido LB agar + ampicillina, aggiungendo 25µl di soluzione di ampicillina
(50mg/ml) per ogni 25 ml di terreno prima di versarlo nelle piastre Petri.
ü
Spatolare su piastre di LB +
ampicillina (50µg/ml) e di LB le quantitŕ di cellule riportate in tabella [Fig.
2]:
|
|
TOP10 + pUC19 |
TOP10 |
|
LB + ampicillina |
10 µl -
100 µl |
10 µl |
|
LB |
|
10 µl |
ü
Incubare le piastre a 37°C per 12-15
ore.
ü
Osservare le piastre ottenute e
prelevare le colonie di E. coli
trasformate [Fig. 3].

Fig. 1:
cellule sottoposte a shock termico durante la trasformazione.
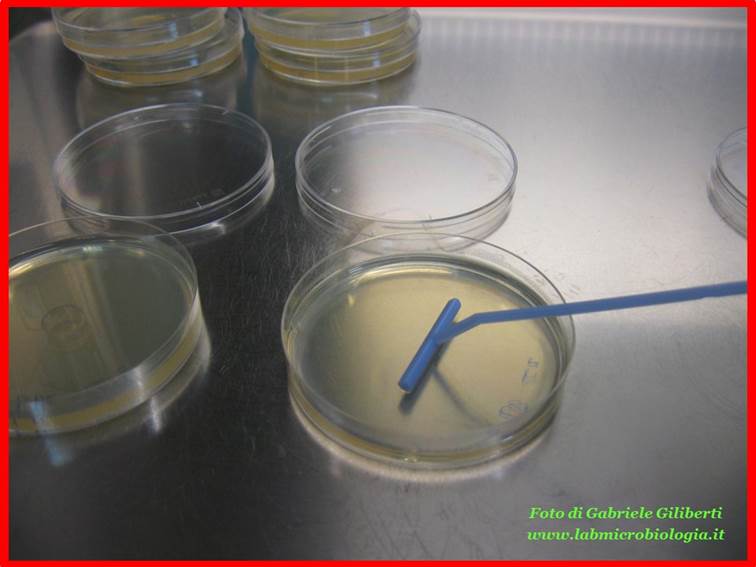
Fig. 2:
spatolamento su piastra delle cellule trasformate.
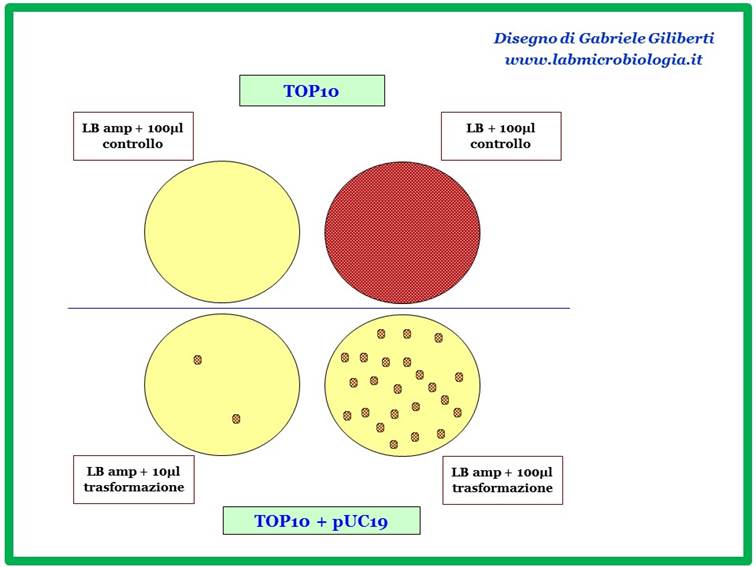
Fig. 3:
schema del risultato atteso dalla trasformazione.



