Laboratorio di
Microbiologia Generale
a cura di Gabriele Giliberti

Identificazione con Api
L’analisi delle caratteristiche biochimiche e fisiologiche
rappresenta un aspetto molto importante nello studio e nella identificazione dei microrganismi. I
test biochimici che permettono di valutare le capacità fermentative, o
metaboliche in generale, e la presenza di attività enzimatiche specifiche hanno
come limitazione principale il tempo occorrente per apprezzare le modificazioni
prodotte dai microrganismi. La semplicità e la riproducibilità, se accoppiate
alla rapidità, consentono invece un ampio uso di questi test; sono così nati
una serie di sistemi miniaturizzati,
forniti da diverse case produttrici, che hanno il vantaggio di evidenziare
differenti attività biochimiche in un’unica soluzione, dal momento che possono
essere inoculati tutti in una volta e letti in un tempo breve (circa 24h). Le
precedenti fasi di identificazione del microrganismo quali isolamento in
terreni selettivi, valutazione delle caratteristiche morfologiche e tintoriali
e test preliminari di orientamento, permettono di individuare il tipo di kit
più appropriato per l’identificazione finale. La galleria Api20E ad esempio è indicata nell’identificazione degli
Enterobatteri; è composta da 20 microprovette contenenti substrati disidratati
che vengono inoculate con una sospensione batterica, ricostituendo così i
terreni. Le reazioni prodotte durante il periodo di incubazione si traducono in
viraggi colorati spontanei o rivelati in seguito all’aggiunta di reattivi. La
lettura di queste reazioni (positiva/negativa) viene registrata su una apposita
tabella che permette di ottenere un profilo numerico; tale profilo numerico
permette l’identificazione della specie attraverso il confronto con un software
di identificazione.
I test biochimici presenti nell’API20E sono:
• [GLU, MAN, INO, SOR, RHA, SAC, MEL, AMY, ARA] Reazioni di fermentazione di diversi zuccheri: la positività
dei test è indicata dal viraggio dell’indicatore da blu a giallo causato dalla
produzione di acidi nel mezzo.
• [CIT] Utilizzazione
del citrato come unica fonte di C: la positività dei test è indicata dal
viraggio dell’indicatore blu di bromofenolo causato dall’alcalinizzazione del
mezzo.
• [VP] Test di
Voges-Proskauer per evidenziare la produzione di acetoina durante la
fermentazione: la positività del test è indicata dallo sviluppo di una
colorazione rossastra, in seguito alla reazione tra acetoina e reagenti
specifici, aggiunti dopo l’incubazione.
• [ONPG] Presenza dell’enzima
β-galattosidasi: la positività del test è indicata dalla colorazione
gialla conferita al terreno dall’ortonitrofenolo, derivato dalla scissione
dell’ortonitrofenil-galattoside operata dall’enzima.
• [ADH] Presenza
dell’enzima arginina deidrolasi: la positività del test è indicata dal viraggio
dell’indicatore a rosso-arancio, causato dall’alcalinizzazione del terreno in
seguito alla trasformazione dell’arginina in citrullina, un’amina alcalina,
operata dall’enzima.
• [LDC, ODC] Presenza degli enzimi lisina e
ornitina decarbossilasi: la positività del test è indicata dal viraggio
dell’indicatore a rosso-arancio, causato dall’alcalinizzazione del terreno in
seguito formazione di amine alcaline causata dagli enzimi.
• [URE] Presenza
dell’enzima idrolitico ureasi: la positività del test è indicata dal viraggio
dell’indicatore a rosso-arancio, causato dall’alcalinizzazione del terreno in
seguito alla idrolisi dell’urea in NH3 e CO2, operata
dall’enzima.
• [GEL] Presenza
dell’enzima proteolitico gelatinasi: la positività del test è indicata dal
fatto che la gelatina se idrolizzata dalle proteasi diventa liquida,
permettendo la diffusione di un pigmento nero che colora la provetta.
• [TDA] Presenza
dell’enzima triptofano deaminasi: la positività del test è indicata dalla
colorazione rossastra che si origina dalla reazione operata dall’enzima in
presenza di triptofano e cloruro di ferro, che viene successivamente aggiunto
dopo l’incubazione.
• [IND] Capacità di
produrre indolo dalla degradazione del triptofano: la positività del test è
indicata dallo sviluppo della colorazione rossastra, in seguito alla reazione
dell’indolo con il reattivo di Kovacs che viene successivamente aggiunto dopo
l’incubazione.
• [H2S]
Produzione di idrogeno solforato: la positività del test è indicata dal fatto
che l’H2S prodotto durante il metabolismo reagisce con il citrato
ferrico formando solfuro ferrico, che forma una precipitato di colore nero.
Per l’esercitazione vengono utilizzati quattro diversi batteri
che permettono di osservare reazioni positive/negative in tutte la provette. Tre
di loro sono enterobatteri (Escherichia
coli, Pseudomonas aeruginosa e Klebsiella pneumoniae) e possono essere
quindi identificati con la Api20E, uno invece, Bacillus subtilis, darà luogo ad un codice non valido per questo
sistema identificativo. I nomi dei microrganismi possono essere mantenuti
eventualmente segreti, se si vuole simulare l’identificazione. A causa della
potenziale patogenicità di alcuni batteri utilizzati, come già discusso, l’apertura
delle piastre e la preparazione delle gallerie vanno eseguite sotto una cappa
di sicurezza biologica a flusso verticale.
Protocollo sperimentale
ü
Preparare 4 vaschette di incubazione
distribuendo in ciascuna circa 5 ml di acqua distillata negli alveoli per
creare un ambiente umido.
ü
Estrarre le gallerie dalla
confezione e metterle nelle vaschette di incubazione [Fig. 1].
ü
Risospendere nell’apposito mezzo una
colonia prelevata da terreno agarizzato.
ü
Utilizzare i seguenti microrganismi:
1) Escherichia coli; 2) Pseudomonas aeruginosa; 3) Bacillus subtilis; 4) Klebsiella pneumoniae.
ü
Riempire provette (parte chiusa) e
cupole (parte aperta) dei test CIT, VP, e GEL con la sospensione batterica.
ü
Riempire solo le provette e non le
cupole degli altri test con la sospensione batterica.
ü
Creare un’anaerobiosi nei test ADH,
LDC, ODC, URE, H2S, ricoprendo le cupole con olio di paraffina [Fig.
2].
ü
Richiudere le vaschette di
incubazione e incubare a 37°C per 12-15 ore.
ü
Osservare le reazioni avvenute nelle
provette GLU, MAN, INO, SOR, RHA, SAC, MEL, AMY, ARA.
ü
Annotare per ciascuna di esse l’esito
positivo [giallo] o negativo [blu/verde] della reazione [Fig. 3].
ü
Osservare la reazione avvenuta nella
provetta ONPG. Annotare l’esito positivo [giallo] o negativo [bianco] della
reazione.
ü
Osservare la reazione avvenuta nella
provetta ADH. Annotare l’esito positivo [rosso/arancio] o negativo [giallo]
della reazione.
ü
Osservare le reazioni avvenute nelle
provette LDC, ODC. Annotare l’esito positivo [rosso/arancio] o negativo
[giallo] delle reazioni.
ü
Osservare la reazione avvenuta nella
provetta CIT. Annotare l’esito positivo [blu] o negativo [verde] della
reazione.
ü
Osservare la reazione avvenuta nella
provetta H2S. Annotare l’esito positivo [nero] o negativo [bianco] della
reazione.
ü
Osservare la reazione avvenuta nella
provetta URE. Annotare l’esito positivo [rosso/arancio] o negativo [giallo]
della reazione.
ü
Osservare la reazione avvenuta nella
provetta GEL. Annotare l’esito positivo [nero] o negativo [bianco] della reazione.
ü
Completare il test nella provetta
TDA, aggiungendo una goccia di reattivo (percloruro di ferro + acqua).
ü
Annotare l’esito positivo
[marrone-rossastro] o negativo [giallo] della reazione.
ü
Completare il test nella provetta
VP, aggiungendo una goccia dei reattivi VP1 (idrossido di potassio + acqua) e
VP2 (α-naftolo + etanolo).
ü
Attendere almeno 10 minuti e
annotare l’esito positivo [rosso intenso] o negativo [bianco/rosa] della
reazione.
ü
Completare il test nella provetta
IND, aggiungendo una goccia di reattivo di James.
ü
Annotare l’esito positivo [rosso] o
negativo [bianco] della reazione.
ü
Confrontare le diverse gallerie
preparate verificando le differenze colorimetriche dei diversi test.
ü
E. coli (1) risulta positivo per LDC,
IND, GLU, MAN, SOR, RHA, MEL, ARA.
ü
P. aeruginosa (2) risulta
positivo per ADH, CIT, TDA, GEL, GLU, ARA.
ü
B. subtilis (3) risulta positivo per ONPG,
VP, GEL.
ü
K. pneumoniae (4) risulta
positivo per LDC, CIT, URE, GLU, MAN, INO, SOR, RHA, SAC, MEL, AMY, ARA.
ü
Eventualmente dai risultati annotati
è possibile ricavare per ciascun campione il profilo numerico a 7 cifre e
confrontarlo con la lista dei profili disponibile [Fig. 4].
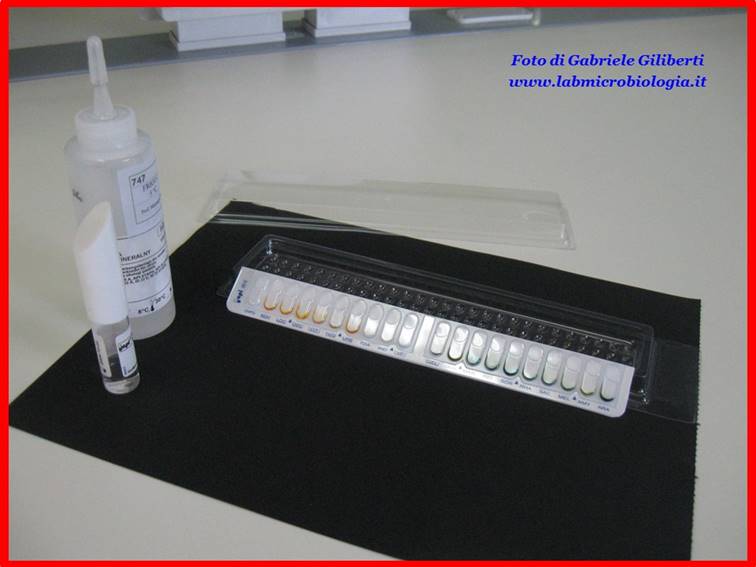
Fig. 1: Galleria
Api20E con terreni liofilizzati pronta per gli inoculi.
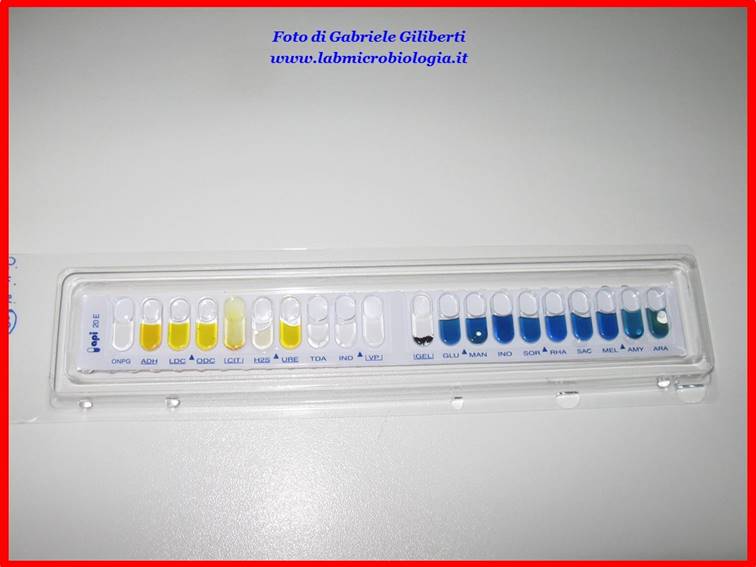
Fig. 2: Galleria
Api20E come appare dopo l’aggiunta della sospensione batterica nelle provette.

Fig. 3: Gallerie
Api20E inoculate con i diversi microrganismi, dopo incubazione a 37°C.
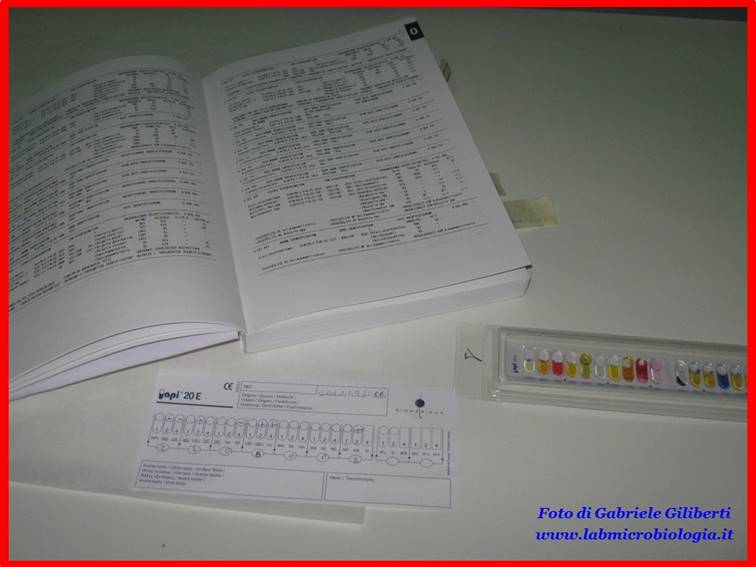
Fig. 4: Scheda
riassuntiva per ottenere il profilo numerico a 7 cifre per l’identificazione
dei batteri.



